«`html
DynamicBind: Глубокий подход к динамическому докингу белка-лиганда и поиску лекарств
Пока значительные успехи были достигнуты в предсказании статических структур белков, понимание динамики белков, влияемой лигандами, является важным для понимания их функции и продвижения открытия лекарств. Традиционные методы докинга часто рассматривают белки как жесткие, что ограничивает их точность. Хотя молекулярно-динамические симуляции могут предложить соответствующие конформации белков, они требуют больших вычислительных ресурсов. Недавние достижения, такие как AlphaFold, предсказывают структуры из последовательностей, но генерируют только несколько конформаций, упуская динамическую природу белков. Это ограничение влияет на точность докинга, поскольку предсказанные AlphaFold-структуры могут не отражать оптимальные конфигурации для связывания лигандов, что приводит к неточным прогнозам.
Практические решения и ценность:
Исследователи из Galixir Technologies, Школы фармацевтических наук Университета Сунь Ятсена, Центра теоретической биологической физики и Факультета химии Райсского университета, а также Глобального института будущих технологий Университета Шанхайского Цзяотун разработали DynamicBind — метод глубокого обучения, использующий эквивариантные геометрические сети диффузии для создания плавного энергетического ландшафта, обеспечивающего эффективные переходы между различными равновесными состояниями. DynamicBind точно предсказывает конформации, специфичные для лигандов, из непривязанных структур белков без голоструктур или обширного сэмплирования. Он превосходит в бенчмарках докинга и виртуального скрининга, учитывая значительные изменения конформации белков и выявляя скрытые карманы в новых целях белков. Этот метод обещает ускорить разработку малых молекул для ранее недоступных целей, продвигая вычислительное открытие лекарств.
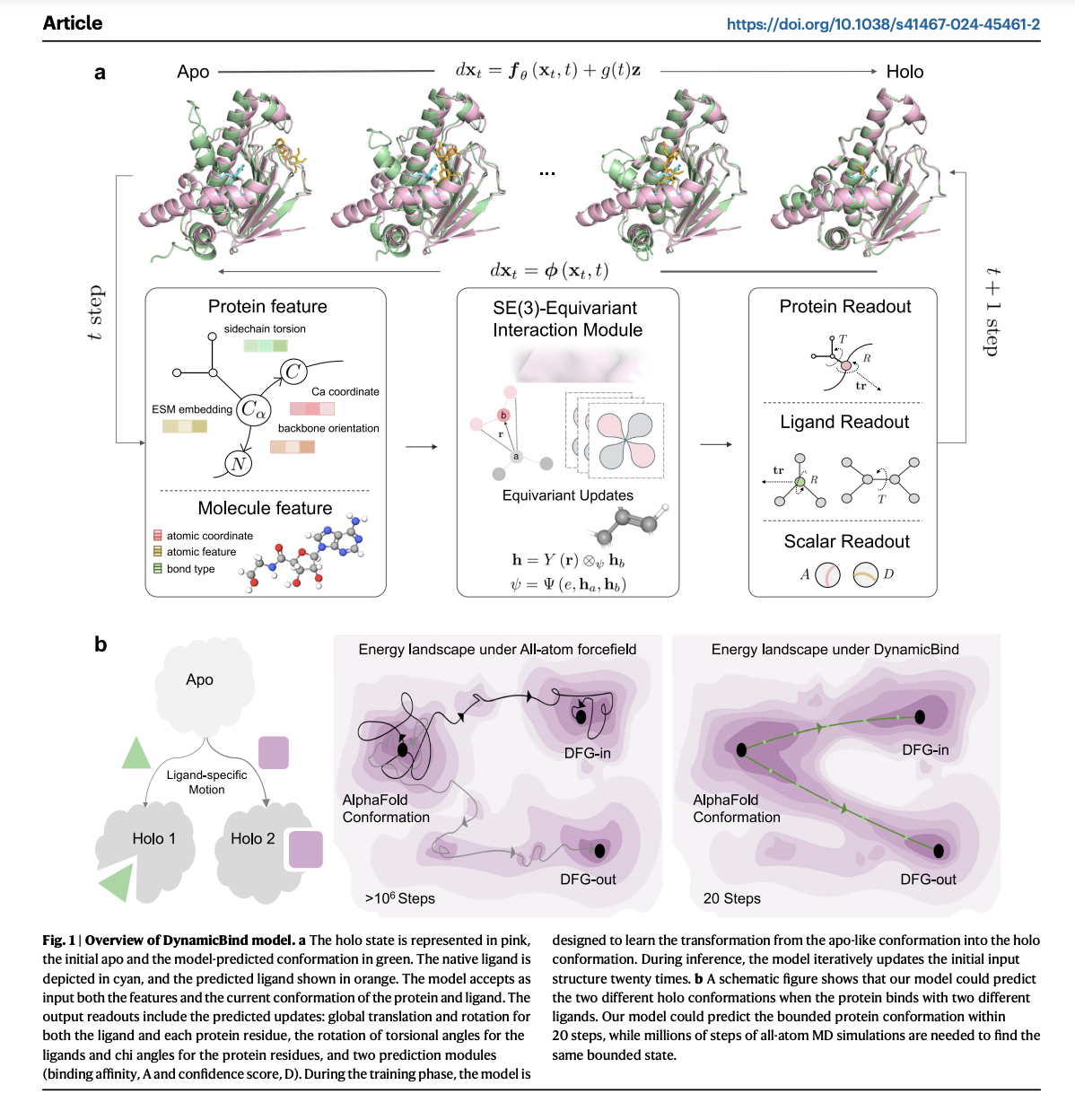
DynamicBind, геометрическая глубокая генеративная модель для динамического докинга, эффективно корректирует конформации белков из начальных предсказаний AlphaFold до состояний, подобных голоструктурам. Он обрабатывает значительные конформационные изменения, такие как переход из состояния DFG-in в DFG-out в киназах, лучше, чем традиционные молекулярно-динамические симуляции. DynamicBind достигает этого, изучая энергетический ландшафт, который минимизирует фрустрацию во время переходов между биологически значимыми состояниями. В отличие от традиционных генераторов Больцмана, DynamicBind обобщаем на новые белки и лиганды.
Модель DynamicBind — это E(3)-эквивариантная, диффузионная графовая нейронная сеть, использующая грубозернистое представление для предсказания конформаций связывания белка-лиганда. Он эффективно преобразует входные структуры, учитывая 3D трансляционные и поворотные изменения, превосходя традиционные методы с меньшим количеством данных. Модель использует морфоподобное преобразование для обучения, интерполируя между кристаллическими и AlphaFold структурами. Используя графовое представление, каждый остаток белка и атом лиганда является узлом с различными характеристиками. DynamicBind обновляет эти узлы через тензорные произведения и диффузионные процессы для предсказания боковых цепей, углов торсиона, трансляций и поворотов, улучшая предсказания аффинности связывания.
DynamicBind — универсальный инструмент для предсказания структур комплексов белок-лиганд, способный адаптироваться к значительным конформационным изменениям белков. Во время вывода он постепенно корректирует положения лигандов и внутренние углы на протяжении 20 итераций, а также адаптирует конформации белков, особенно углы боковых цепей, улучшая предсказанные AlphaFold-структуры. В отличие от традиционных моделей, он использует морфоподобное преобразование, а не гауссовские шумовые возмущения, что улучшает способность модели захватывать биологически значимые конформационные изменения. DynamicBind превосходит в предсказании позы лиганда, уменьшая столкновения и выявляя скрытые карманы, как показано на различных бенчмарках и кейс-стади, демонстрируя свой потенциал для приложений в области открытия лекарств.
В заключение, DynamicBind интегрирует генерацию конформации белка и предсказание позы лиганда в единую глубокую систему обучения, значительно быстрее традиционных молекулярно-динамических симуляций. В отличие от обычных методов докинга, требующих заранее определенных связывающих карманов, DynamicBind выполняет глобальный докинг, что идеально подходит для выявления скрытых карманов. Это уменьшает потенциальные побочные эффекты путем нацеливания на конкретные белки и помогает в открытии лекарств путем предсказания непреднамеренных белковых целей или выявления целей в фенотипическом скрининге. Хотя он показывает отличную производительность, требуются улучшения для лучшей обобщения на белки с низкой гомологией последовательностей. Продвижения в области крио-ЭМ и включение данных об аффинности связывания могут улучшить возможности DynamicBind.
Проверьте статью и GitHub. Вся заслуга за это исследование принадлежит исследователям этого проекта. Также не забудьте подписаться на нас в Twitter. Присоединяйтесь к нашему каналу в Telegram, Discord и группе в LinkedIn.
Если вам нравится наша работа, вам понравится наш новостной бюллетень.
Не забудьте присоединиться к нашему SubReddit с 42 тысячами подписчиков.
Статья опубликована на портале MarkTechPost.
«`