«`html
Новые возможности и вызовы в прогнозировании специфичности TCR: от кластеризации до моделей языка белков
Недавние достижения в иммунном секвенировании и экспериментальных методах порождают обширные данные репертуара Т-клеточных рецепторов (TCR), позволяя создавать модели для прогнозирования специфичности связывания TCR. T-клетки играют роль в адаптивной иммунной системе, оркестрируя целенаправленные иммунные ответы через TCR, распознающие незарегистрированные антигены от патогенов или больных клеток. Разнообразие TCR, необходимое для распознавания разнообразных антигенов, порождается через случайное перестройство ДНК, включающее сегменты генов V, D и J. В то время как теоретическое разнообразие TCR чрезвычайно высоко, фактическое разнообразие у индивида гораздо меньше. TCR взаимодействуют с пептидами на основном комплексе гистосовместимости (pMHC), причем некоторые TCR распознают многочисленные комплексы pMHC.
Прогресс в моделях прогнозирования специфичности TCR
Исследователи из IBM Research Europe, Института вычислительных наук о жизни при Зюрихском университете прикладных наук и медицинской школы Йель рассматривают эволюцию вычислительных моделей для прогнозирования специфичности связывания TCR. С упором на машинное обучение, они охватывают ранние подходы кластеризации без учителя, обученные модели и трансформационное воздействие языковых моделей белков (PLM) в биоинформатике, особенно в анализе специфичности TCR. Обзор затрагивает предвзятость набора данных, проблемы обобщения и недостатки валидации моделей. Он подчеркивает важность улучшения интерпретируемости моделей и извлечения биологических идей из больших, сложных моделей для улучшения прогнозирования связывания TCR-pMHC и революционизации разработки иммунотерапии.
Данные о специфичности TCR поступают из баз данных, таких как VDJdb и McPas-TCR, но у этих наборов данных есть существенные ограничения. Массовое секвенирование является высокопроизводительным и экономически эффективным, но не может обнаружить сопряженные α и β цепи, в то время как дорогостоящие технологии одиночных клеток, способные это делать, недостаточно представлены. Большинство наборов данных фокусируются на ограниченном количестве эпитопов, в основном вирусного происхождения и связанных с общими аллелями HLA, что показывает значительную предвзятость. Кроме того, отсутствие отрицательных данных усложняет разработку обученных моделей машинного обучения. Создание искусственных отрицательных пар вносит предвзятость, а высокопроизводительные модели могут запоминать последовательности, приводя к излишне оптимистичным результатам. Обеспечение того, чтобы сгенерированные отрицательные пары точно отражали истинные небиндивные распределения, остается вызовом.
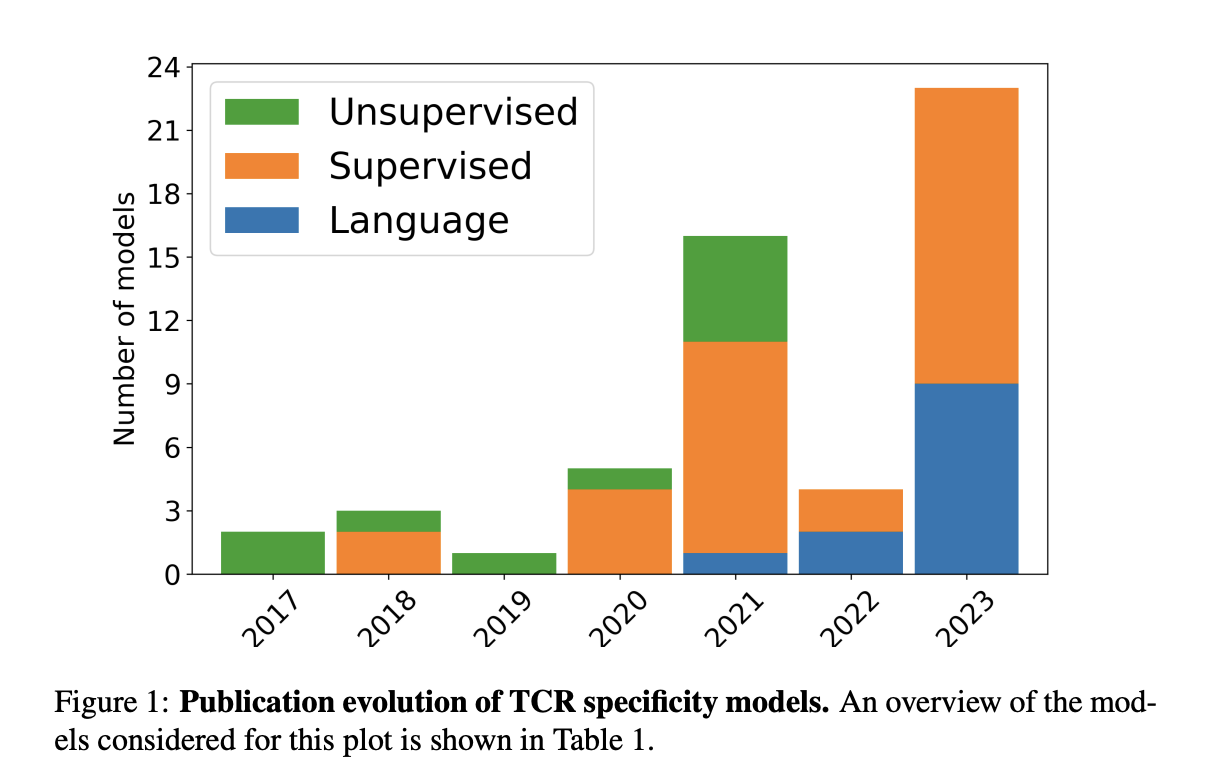
С 2017 года моделирование специфичности TCR значительно эволюционировало, начиная с методов кластеризации без учителя. Начальные модели, такие как TCRdist и GLIPH, группировали TCR на основе сходства последовательностей и биохимических свойств. Эти методы продемонстрировали, что последовательности TCR содержат ценную информацию о специфичности, но столкнулись с сложными нелинейными взаимодействиями. Это послужило толчком к разработке обученных моделей, использующих техники машинного обучения для более эффективной обработки увеличивающейся сложности данных. Ранние обученные модели, включая TCRGP и TCRex, использовали классификаторы, такие как гауссовы процессы и случайные леса, для прогнозирования специфичности TCR. Тем временем подходы на основе нейронных сетей, такие как NetTCR и DeepTCR, использовали передовые архитектуры для улучшения предсказательной точности.
Внедрение языковых моделей белков (PLM) стало последним достижением в прогнозировании специфичности TCR. На основе архитектур Transformer, эти модели были обучены на обширных наборах данных последовательностей белков и достигли замечательной производительности в различных задачах, связанных с белками. Например, TCR-BERT и STAPLER использовали модели на основе BERT, настроенные на классификацию TCR и антигенов, демонстрируя эффективность PLM в улавливании сложных последовательностных взаимодействий. Несмотря на их успех, остаются вызовы в устранении лексической неоднозначности и улучшении интерпретируемости моделей. Будущие улучшения в оптимизации встраивания и адаптации методов интерпретируемости, специфических для последовательностей белков, критичны для дальнейших успехов в прогнозировании специфичности TCR.
Точное прогнозирование специфичности TCR необходимо для улучшения иммунотерапий и понимания аутоиммунных заболеваний. Ограниченные и предвзятые данные, особенно информация об эпитопах, ставят под сомнение текущие модели, затрудняя обобщение на новые эпитопы. Прогресс в машинном обучении, включая сверточные нейронные сети, рекуррентные нейронные сети, обучение передачи и PLM, значительно улучшил модели прогнозирования TCR, но остаются вызовы, особенно в прогнозировании специфичности для новых эпитопов. Бенчмарки, такие как IMMREP22 и IMMREP23, подчеркивают трудности в справедливом сравнении моделей и их обобщении. Адаптация моделей TCR для прогнозирования BCR, который включает нелинейные эпитопы и сложные взаимодействия с антигенами, представляет дополнительные вычислительные вызовы.
Проверьте статью. Вся заслуга за это исследование принадлежит исследователям этого проекта. Также не забудьте подписаться на наш Twitter и присоединиться к нашему каналу в Telegram и группе в LinkedIn. Если вам нравится наша работа, вам понравится наша рассылка.
Не забудьте присоединиться к нашему сообществу в Reddit
Найдите предстоящие вебинары по ИИ здесь
Статья опубликована на сайте MarkTechPost.
«`






















